- 封面故事
- 2022年
- 628期-生物資訊大解碼(4月號)
文章專區
2022-04-01以「次世代定序」完成精準醫療,更能協助生命科學領域的探索
628 期
Author 作者
趙盈婷/中央大學生醫科學與工程學系系統生物與生物資訊博士候選人。許藝瓊/中央大學生醫科學與工程學系副教授。
Take Home Message
◆ 1977 年,英國生物化學家桑格發明了第一代核酸定序技術「鏈終止法」。他利用雙去氧三磷酸核苷酸(ddNTP)終止DNA合成,再藉由分析終止的產物片段反推其序列,使用方法簡單、快速,且反應成本較低。
◆ 2005 年,次世代定序技術(NGS)被推出,使科學家能同時定序大量DNA,進而提升通量、降低成本、增加速度,也讓NGS正式用於精準醫學,更適用於與生命科學相關領域的DNA探索。
◆ 透過NGS的定序技術,我們能解碼人類的全基因組,或是建立一個全面性的人類微生物體學資料庫,更可以分析環境中的微生物組成。
去氧核糖核酸(DNA)是生物體中重要的遺傳物質,當中隱藏的各種遺傳密碼,令許多研究者既興奮又期待,只想一窺這複雜的生物奧祕。於是在1990 年美國發起了人類基因體計畫(Human Genome Project, HGP),更在2003 年宣告完成完整人類基因組解碼。不過要完成這些艱困的任務,有個先決技術必須克服——該怎麼解密DNA?我們要如何知道DNA的序列?
第一代核酸定序技術的誕生
1953年,美國分子生物學家華生(James Watson)與英國生物學家克里克(Francis Crick)破解了DNA的結構,也讓生物學研究從巨觀的生物現象及以外觀表徵判斷疾病或種間差異,轉而以微觀的分子生物取而代之。到了1977 年,英國生物化學家桑格(Frederick Sanger)發明了名為「鏈終止法」(chain-termination method)的第一代核酸定序技術,使得核酸定序成為生物技術發展的重要基礎,桑格也因此榮獲1980 年的諾貝爾化學獎。
此DNA定序技術的原理是在DNA合成的過程中,其原料為去氧三磷酸核苷酸(deoxyribonucleotide,亦稱dNTP, 如dATP、dGTP、dCTP、dTTP),若是能利用合成DNA 原料的差異,將原本的去氧三磷酸核苷酸改變為「雙去氧三磷酸核苷酸」(dideoxynucleotide, 亦稱ddNTP, 如ddATP、ddGTP、ddCTP、ddTTP),進而終止DNA 繼續合成的過程,就能藉由分析終止的產物片段,反推其序列(圖一)。桑格發明的定序方法在現代稱為第一代定序,為分子生物學領域當中最基本的DNA解碼工具,使用方法簡單快速且反應成本較低。
圖一|桑格定序的原理
更快、更有效率、成本更低的「次世代定序」
隨著定序技術的演進,美國在1990年發起了人類基因組計畫。第一代定序技術在通量、大量定序所耗費的成本及速度上,已無法滿足日益增長的大規模全基因組定序需求。因此在2005年,次世代定序技術(next generation sequencing, NGS)被推出,由於此技術可同時定序大量DNA,進而提升通量、降低成本、增加速度。
早期市面上的NGS定序儀有Illumina、ABI、Roche這3家廠牌,不過隨著時間的演進,目前科學期刊當中,約70%以上採用Illumina平臺作為定序工具。Illumina的定序技術主要是利用大量的DNA短片段定序序列,利用物理性方式將DNA打斷成大約300 ∼ 500個鹼基的小片段,並在兩端接上轉接子(adaptor),再把含有轉接子的序列,鋪到上方有轉接子互補序列的晶片上,透過橋式聚合酶鏈鎖反應(bridge PCR)進行擴增。再經由標記不同鹼基的特定序列,移除螢光分子的dNTP與反應試劑,重覆進行螢光標記移除與偵測,達到快速且大量的定序結果(圖二)。最後再用生物資訊技術分析資料,判別出每一條序列的正確鹼基對。
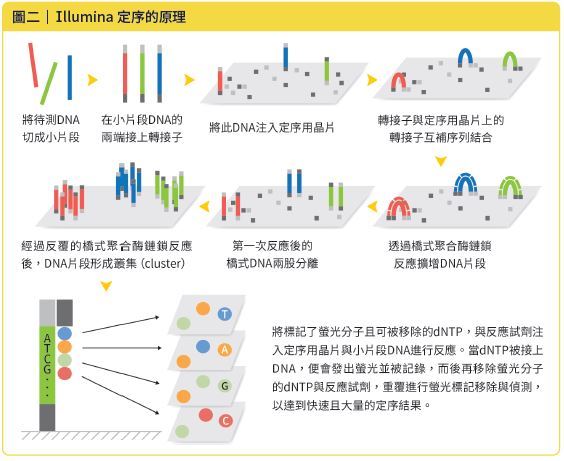
圖二| Illumina 定序的原理
由於NGS 的快速發展,根據美國國家衛生研究院(National Institutes of Health, NIH)統計,近20年來每百萬鹼基定序的成本已從5000美元大幅降至0.01美元;人類全基因體定序成本也從一個人近億美元降至1000美元以下。使得NGS成為不僅用於精準醫學的重要技術,甚至還能用於生命科學相關領域的DNA探索。
而另一個應用的範疇,則是透過NGS研究特定環境下微生物的組成,與其相對數量。由於微生物是一種難以用肉眼直接看到的微小生物,早期需要先藉由人工培養,再以顯微鏡來觀察進行研究。過去,估計只有約1%的環境細菌能夠在實驗室環境下培養,近來也因技術上的經驗積累,把可培養的細菌種類提高至50%。……【更多內容請閱讀科學月刊第628期】