- 科技報導
文章專區
2020-03-11追捕新型冠狀病毒的福爾摩斯?談張鋒的SHERLOCK試紙型檢測法
459 期
Author 作者
黃子懿/臺灣大學醫學系一年級生;陳佑宗/臺灣大學基因體暨蛋白體醫學研究所副教授。
近日新型冠狀病毒(SARS-CoV-2)疫情持續延燒,相關各界研究更是如火如荼地進行。今(2020)年2月14日,美國麻省理工學院(Massachusetts Institute of Technology, MIT)張鋒等人宣布,他們與博德研究所(Broad Instituteof MIT and Harvard)、麥戈文腦科學研究所(McGovern Institute forBrain Research, MIT)等機構,將一種運用CRISPR/Cas13a、名為「SHERLOCK」的試紙型快速檢測法應用在SARS-CoV-2引起的2019冠狀病毒疾病(COVID-19)篩檢上,並能在一小時內得知樣本中是否存在病毒RNA,被視為是未來檢疫的重要研究及發展方向(Zhang F et al., submitted,圖一)。本文將介紹這項取名源自小說偵探人物夏洛克.福爾摩斯(Sherlock Holmes)的新興技術。
回到源頭-細菌的CRISPR/Cas
想瞭解SHERLOCK的操作原理,得先知道該檢測法所使用的技術──CRISPR/Cas系統。
CRISPR的全名為Clustered Regularly Interspaced Short Palindromic Repeats(群聚且有規律間隔的短回文重複序列),是在細菌DNA中發現的一系列重複序列,可視為細菌的一種免疫機制。當細菌遭噬菌體(一種病毒)感染時,便會切取一小段噬菌體的DNA,然後儲存於CRISPR之間的間隔序列(spacer),以便未來再次遭到感染時,能更快識別並摧毀同種噬菌體。而Cas指的是由CRISPR相關基因(CRISPR associated gene,簡稱cas基因)所編碼的蛋白質酵素,會受引導RNA(guide RNA, gRNA)領導跟特定核酸序列結合,專一性相當高,功能包含解開DNA雙螺旋、切割DNA和RNA分子等(圖二)。科學家們利用細菌這一套獨特的免疫機制,設計出一系列能修改核酸分子的工具。
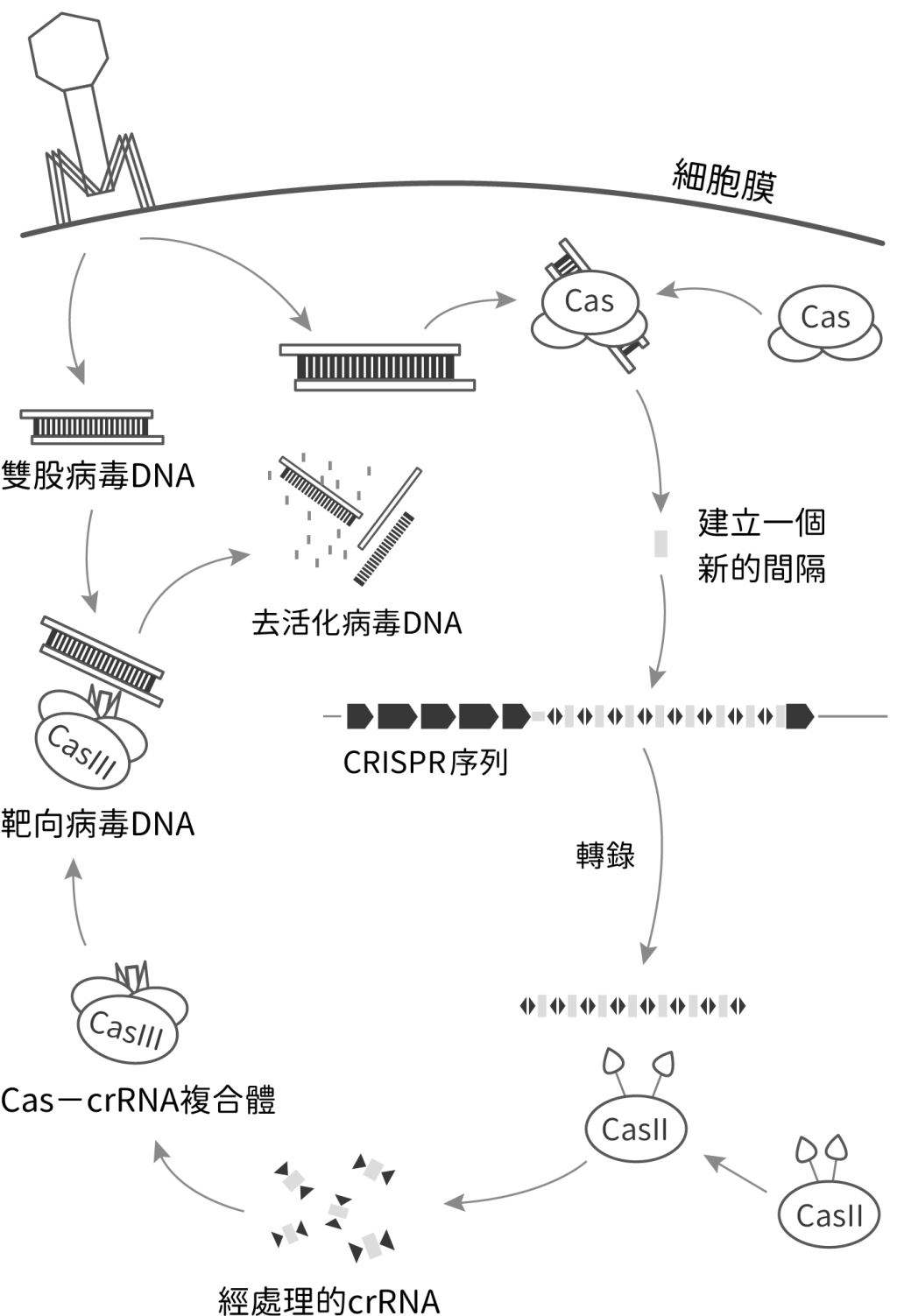
圖二:CRISPR的可能機制示意圖。
(屬於第二類、第二型)。而SHERLOCK這個技術的主角則是CRISPR/Cas13(屬於第二類、第六型),與CRISPR/Cas9的差異在於:前者結合並切割RNA,後者則是針對DNA。
福爾摩斯登場-解決基因檢測的靈敏度問題
SHERLOCK是張鋒和團隊在2017年4月發表於Science期刊的CRISPR檢驗方式,全名為Specific High-sensitivity Enzymatic Reporter unLOCKing(特異性高靈敏度酶促報導受質解鎖)(Gootenberg JS et al., 2017)。其中所用的Cas13a蛋白質(LwaCas13a)來自韋德纖毛菌(Leptotrichia wadei,暫譯),具有特殊的「附帶切割(collateral cleavage)性質」。從下頁圖三中可以看到,當Cas13a經gRNA引導跟目標RNA結合後,會無差別地切割周遭所有的RNA分子,無論它們是否為目標序列(target sequence)。
張鋒等人利用Cas13a的這個特性,結合設計好的、針對特定病原基因體序列的gRNA,並搭配一種經特殊設計、被切割後會發出螢光的報導RNA(reporter RNA),驗證了SHERLOCK能偵測病毒RNA,且靈敏度可達50飛莫耳每升(femtomolar)等級(1fM=10-15M)。儘管這樣的靈敏度已較過去其它Cas13蛋白質來的高,但臨床診斷應用需要達到阿莫耳每升(attomolar)等級(1 aM=10-18M),約等於每微升(μL)樣本中僅含1個核酸序列分子即可被偵測)的等級。於是他們嘗試結合許多不同的等溫放大法(isothermal amplification),最後發現重組酶聚合酶放大(recombinase polymerase amplification, RPA)是最適合的,能夠讓靈敏度達到這個等級。
RPA是利用已經設計好的引子(primer)和目標序列鹼基配對,再搭配重組酶以及聚合酶大量擴增原樣本中的核酸含量。相較於需要反覆升溫、降溫的聚合酶連鎖反應(polymerase chain reaction, PCR),RPA可以在37~42℃間等溫反應,因此不需要熱循環機(thermocycler),使用上更為便捷。不過須注意的是,RPA最終產物為DNA,所以尚需經來自T7噬菌體的T7 RNA聚合酶轉錄成RNA,才能與Cas13a反應(圖四)。
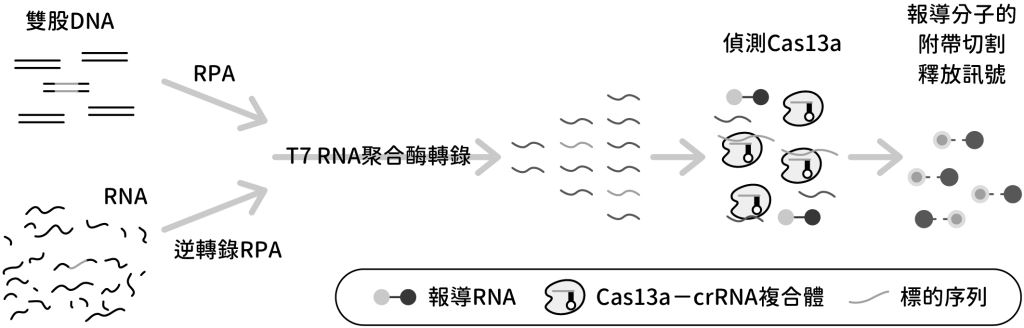
圖四:SHERLOCK 的基本流程。
多種病毒檢測,都難不倒它
找到最佳方法後, 研究團隊便合成茲卡病毒(zika virus, ZIKV)和登革熱病毒(flavivirus dengue, DENV)的RNA,將它們包覆於慢病毒(lentivirus)中測試SHERLOCK。結果顯示,SHERLOCK能偵測到濃度僅2阿莫耳每升的病毒RNA,且能區別過去因症狀相似而常無法分明的茲卡病毒和登革熱病毒。同時,SHERLOCK也能檢測出病人血清、尿液或唾液中的茲卡病毒,為其未來臨床應用奠下良好基礎。
到了2018年4月,張鋒的研究團隊更進一步發表改良後的技術,結合濾紙層析(lateral flow),使未被切割的報導RNA可附著於試紙上的底線,被切割的部分則附著於試紙上方。如此一來,SHERLOCK便成為僅需將試紙浸入經RPA處理、Cas13a反應後的樣本就能知道結果的試劑組,如同驗孕棒一般簡單,過程不須使用昂貴的設備。此外,原本一次只能針對一種目標,改良後最多可以同時偵測四種目標序列,因此能減少樣本取量。該檢驗法除了可偵測傳染性病毒,也擴展至檢驗致癌的基因變異、識別某些具抗生素耐藥性細菌的基因等。
一小時追捕兇手-SHERLOCK三步驟檢測COVID-19
不過讀者應該更想知道,該試劑如何用在此疾病的篩檢上吧!
目前檢驗冠狀病毒,主要利用反轉錄聚合酶連鎖反應(簡稱RTPCR),過程至少需要四小時,且僅大型醫院具備相關儀器,使得醫療體系面對大量要求篩檢的民眾時容易無法負荷。
有鑑於上述,張鋒團隊試圖將SHERLOCK應用於此。他們從SARS-CoV-2的基因體中,挑選了S基因和Orf1ab基因作為目標序列,並設計相對應的gRNA以及RPA所需的引子。同時,他們合成這兩個基因的RNA片段,再將它做連續稀釋(serial dilution)以進行後續試驗。根據團隊公布的操作方法(Zhang F et al., submitted),利用SHERLOCK檢測COVID-19僅需三步驟。
事前準備:將檢體經過核酸萃取處理(nucleic acid extraction)。此次實驗並非以病人檢體試驗,而是以稀釋的合成病毒RNA片段,所以無需萃取。
1. 等溫放大(約25分鐘):利用前述的RPA法提升檢體中的核酸含量。可以使用現成的RPA kit,放在42℃水浴中進行反應。
2. Cas13偵測(約30分鐘):將T7聚合酶、gRNA、LwaCas13a蛋白質和報導RNA放入第一步處理完的溶液中。均勻混合後,置於37℃水浴。
3. 試紙讀取(約2分鐘):用緩衝溶液稀釋反應完的溶液,插入專用試紙讀取結果。
結果分析:試紙底部的控制組底線(control band)應該都要呈色。若有偵測到病毒RNA,則會出現兩條線。從圖五可知,如果檢體對於S基因和Orf1ab基因皆呈現兩條線,則代表COVID-19陽性(positive)。
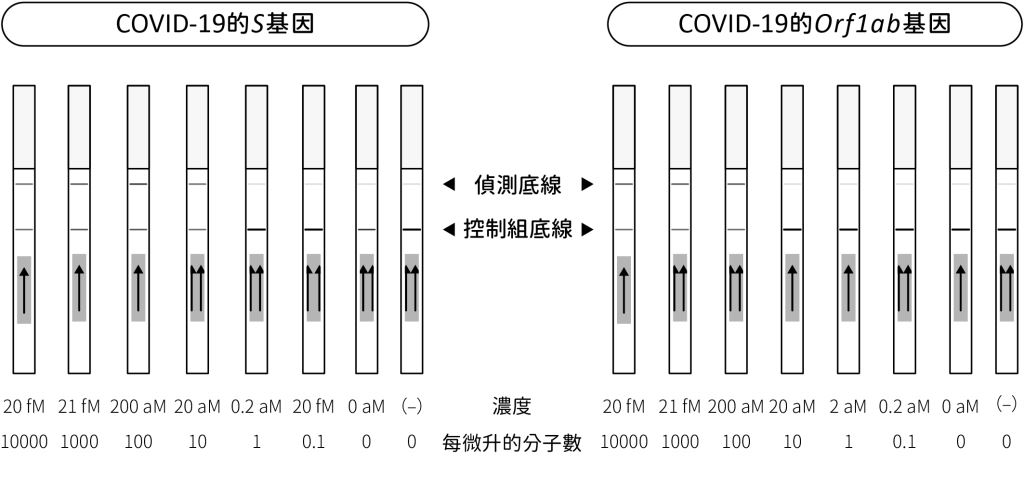
圖五:SHERLOCK 試劑組測試COVID-19之結果示意圖。故事才剛開始-SHERLOCK的未來及挑戰
SHERLOCK相較於傳統的RTPCR在操作上更為簡單、迅速,完全不需要複雜的儀器。若能順利發展,可以讓一般診所即能對COVID-19、登革熱等傳染病進行檢測,減輕大型醫院的負擔,甚至還可以攜帶至醫療資源較匱乏的地區協助控制疫情,如檢疫非洲的伊波拉病毒。
然而,由於SHERLOCK所需的報導RNA及Cas13蛋白價格仍然偏高,如何便宜地大量生產是相當大的挑戰,而成敗的關鍵或許在於:能否使用最少的原料達到足夠的靈敏度及精準度,進而壓低成本。此外,如果要到偏遠地區進行檢疫,如何確保蛋白質及相關試劑在運送過程中不會變質,也會是一大考驗。同時須注意到,若在非最適反應溫度的條件下,RPA是否能維持其專一性?所設計的gRNA是否提供足夠的特異性?所使用的Cas13a是否提供足夠的精準度而不致有脫靶效應(off-target effect)、造成偽陽性(false positive)的檢測結果?這些考量都需要未來以更嚴密的臨床試驗才能進一步確認。
儘管如此,SHERLOCK仍是發展防疫工具的良好研究方向。張鋒的團隊歡迎各界學者與他們聯繫以尋求協助和指導,並願意提供初學者試劑套組(starter kit)以利各方進行測試。期待不久後SHERLOCK能如其名,成為調查基因和病毒序列的大偵探!
參考資料
1. Zhang F, Abudayyeh OO, Gootenberg JS, A protocol for detection of COVID-19 using CRISPR diagnostics (submitted, 2020).
2. KS Makarova, YI Wolf, J Iranzo et al., Evolutionary classification of CRISPR–Cas systems: a burst of class 2 and derived variants, Nature Reviews Microbiology, Vol 18: 67-83, 2020.
3. Gootenberg JS et al., Nucleic acid detection withCRISPR/Cas13a/C2C12, Science, Vol 356: 438-442, 2017.
SHERLOCK相關延伸閱讀
McGovern Institute解釋SHERLOCK的影片(三分鐘)SHERLOCK: A CRISPR Tool to Detect Disease.