- 專欄
文章專區
2025-02-11番茄之敵的秘密武器 隱藏在病毒基因組內的抗病密碼
662 期
Author 作者
劉明容 | 任職於中央研究院,研究為植物及植病毒基因表現機制及其生物意義。林達興 | 慈濟大學生命科學系碩士,現任職於劉明容老師實驗室。
Take Home Message
• 番茄黃化捲葉病是一種嚴重威脅番茄產量的病害,目前主要依靠防治傳播病毒的銀葉粉蝨和移除病株兩種方法防治,但效果有限。
• 研究團隊在番茄黃化捲葉病毒基因組中發現17 個新的轉譯起始點,顯示病毒透過非典型轉譯機制產生更多未知的蛋白質。
• 團隊也重新定義致病基因AV2 的轉譯位置,並發現可能參與病毒複製及移動的BV2 基因。這些發現將有助於了解病毒感染機制,為開發RNA 干擾防治方法和抗病育種提供新方向。
番茄是在全球廣泛種植的經濟作物。根據農業部統計,2021 年臺灣番茄種植面積達4090 公頃,產量共9 萬8130.847 公噸。不過番茄生長過程中有一種十分常見的病害――番茄黃化捲葉病,此病由番茄黃化捲葉病毒(tomato yellow leaf curl virus)所導致,被感染後的植物會出現植株矮化、新葉變小、扭曲變形、黃化、花枯萎等病症。在田間,番茄黃化捲葉病最嚴重時會造成100%的損失,極度影響果實產量。
番茄黃化捲葉病毒的防治困境
番茄黃化捲葉病毒為雙生病毒科豆類金黃嵌紋病毒屬(Begomovirus),具有一或兩條環狀單股DNA。由於此病毒基因變異度大,至今在全球已發現40 多種品系。在臺灣也發現了許多不同的病毒株,它們都會導致番茄黃化捲葉病,其中泰國種與臺灣種的混合品系是造成植物感染的主要品系之一,而泰國種的致病性則最強。
番茄黃化捲葉病尚無有效藥劑可防治。目前在田間主要採取兩種防治對策,一是針對傳播病毒的中間寄主銀葉粉蝨(Bemisia tabaci),二是移除發病植株進行防治。然而粉蝨繁殖速度快、抗藥性高使得防治難以奏效,而移除發病植株的方式又治標不治本。因此了解番茄黃化捲葉病毒的感染和致病機制,在現階段就顯得尤為重要。
巧妙的非典型轉譯機制-在有限基因體中堆疊更多功能蛋白
病毒為了感染寄主並完成複製、增生,必須躲避或抑制寄主的免疫機制,同時還要調控寄主基因表現,為病毒營造出一個適合複製的環境。在此過程中,病毒需要眾多功能各異的蛋白質協助。然而,植物病毒的基因體相對較小,以番茄黃化捲葉病毒為例子,該病毒基因體長度有2600 ~5300 個核苷酸,遠小於宿主番茄的基因體(約900 Mb,1 Mb 代表100 萬個核苷酸長度)。因此,病毒能夠轉譯(translation)製造的蛋白質種類有限,而如何在這細小的基因體中表達多種不同的蛋白質,就成為病毒生存的關鍵。
.jpg)
(AJC1 from UK, CC BY-SA 2.0, Wikimedia Commons)

(Dinkum, CC0, Wikimedia Commons)
在生物體中,蛋白質合成的過程需要藉由訊息RNA(messenger RNA, mRNA)提供遺傳訊息,並由核糖體(ribosome)負責執行合成的任務。mRNA 是由一連串核苷酸組成,包含蛋白質序列的遺傳訊息。核糖體會與mRNA 結合,沿著mRNA 的5' 端往3' 端尾的方向進行掃描,搜尋在mRNA 上的典型起始密碼子AUG。當核糖體搜尋到AUG時,就會開始蛋白質的合成。合成過程中,核糖體會以三個核苷酸為一組密碼子,並為配對相應的胺基酸製造出胜肽(peptide)。直到核糖體遇到終止密碼子(UAG、UGA、UAA)時會停止會胜肽的合成。隨後,胜肽會被折疊和加上化學修飾,成為具有功能的蛋白質。
這些可以轉譯出蛋白質的基因體區域稱為轉譯區間(open reading frame, ORF)。而轉譯區間在過去有嚴格的定義,生物學家認為有效的轉譯區間必須大於100 個胺基酸,且區間之間不可以重疊。以番茄黃化捲葉病毒的泰國種為例,它具有DNA-A 和DNA-B 兩個環狀的DNA,基因體長度都約為2600 個核苷酸。已知DNA-A 上有六個AUG,DNA-B 上有兩個AUG,共具有八個轉譯區間,可轉譯產生出八種蛋白質。
然而有趣的是,研究也發現在mRNA 轉譯產生蛋白質的過程中,核糖體除了可辨認上述提到的AUG,也可以藉由辨認非典型的轉譯起始點(例如CUG、ACG 等起始密碼子)來啟動轉譯機制,開始合成蛋白質。因此,透過調控核糖體與mRNA 結合的區域,核糖體可在同一條mRNA 的不同轉譯起始點開始生產蛋白質,創造出具有不同生理功能的蛋白質(圖一)。這種現象在植物病毒、植物、人類等多種生物都有被觀察到,說明非典型轉譯是廣泛存在於不同物種的轉譯機制。而非典型轉譯也讓病毒得以在有限的基因體中盡可能容納更多不同的轉譯區間,產生更多樣的病毒蛋白質。
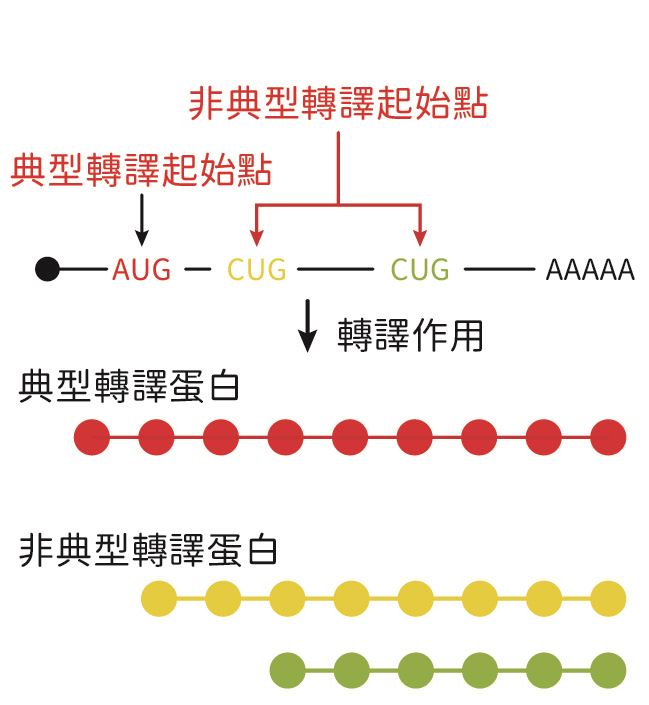
圖一 | 訊息RNA
蛋白質合成可從mRNA上不同的轉譯起始點(包含典型及非典型轉譯起始點)開始進行轉譯,產生出不同的蛋白質(包含典型及非典型蛋白質)。(資料來源:作者提供)
然而,核糖體如何辨認出非典型轉譯起始點和相對應的非典型轉譯區間?目前科學家並不清楚它們在mRNA 上的序列特徵,因此難以預測起始點和轉譯區間的存在,這是了解病毒感染和致病機制的一大阻礙。
揭祕隱藏的轉譯區間
為解決此問題,筆者團隊結合分子生物及電腦分析的概念,採用核糖體分析(ribosome profiling)和轉譯起始抑制劑處理(translation initiation inhibitor treatment)這兩種技術,希望能找出在植物病毒mRNA 序列上的起始密碼子位置及相對應產生出的病毒蛋白質。藉由核糖體分析和轉譯起始抑制劑處理這兩種技術,將正在進行轉譯的核糖體固定在mRNA 上;再進一步用高通量定序(high-throughput sequencing)及大數據分析方法,定位核糖體在mRNA 上位置,以找出在mRNA 序列上、尚未被發現的起始密碼子位置及相對應的轉譯區間,藉此找出病毒基因體中含有的蛋白質訊息。
核糖體分析-捕捉蛋白質合成瞬間
核糖體分析是一種找出mRNA 上轉譯區間的方法,是將正在進行轉譯的核糖體,固定在mRNA上,再進一步 ……【更多內容請閱讀科學月刊第662期】